Minha Avó Paterna Haplogroup mtDNA A2 Nativo Americano
 Mapas do Nativo Americano - mtDNA Haplogroup A2
Mapas do Nativo Americano - mtDNA Haplogroup A2
Minha Avó Paterna Haplogroup mtDNA A2 Nativo Americano
Bem-vindo à
Clã Aiyana
Clã Aiyana
Em genética humana, é um Haplogrupo A DNA Mitocondrial humano (mtDNA) Haplogrupo.
A haplogrupo é acreditado para ter originado na Ásia cerca de 60.000 anos antes do presente. Sua Haplogrupo ancestral era Haplogroup N.
A Haplogrupo é encontrado em toda a Ásia moderna. Seu subgrupo A1 é encontrada no norte e da Ásia Central, enquanto o seu subgrupo A2 é encontrado na Sibéria e é também um dos cinco Haplogrupos encontrados nos povos Indígenas das Américas, sendo os outros B, C, D e X.
A múmia "Juanita", do Peru, também chamado de "Ice Maiden", tem sido demonstrado que pertencem ao Haplogrupo Mitocondrial A.
Em seu famoso livro As Sete Filhas de Eva, Bryan Sykes nomeado o autor deste Haplogrupo Mitocondrial Aiyana.
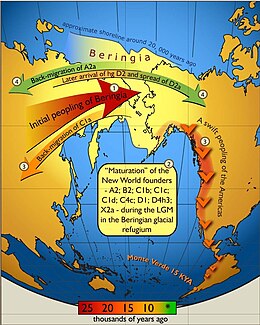
Norte e América do Sul são os continentes mais recentemente colonizado por humanos modernos. A migração modelo a hipótese de que até o final da última Idade do Gelo (cerca de 25.000 anos atrás) um pequeno número de caçadores e coletores seguiu os padrões de migração de animais de grande porte do norte da Ásia para a América do Norte através da ponte de gelo do Estreito de Bering. Essa expansão ocorreu de forma rápida, e empurrou para os climas mais quentes da América Central e do Sul, devido às condições climáticas adversas e de gelo que cobria a maior parte da América do Norte, é provável que pequenos grupos de indivíduos permaneceram espalhados Alasca atual, em seguida, mudou para o sul em uma segunda onda , como as condições climáticas melhoraram gradualmente.
As primeiras quatro letras do alfabeto são usados para designar as quatro principais mtDNA Haplogrupos Nativos Americanos (A, B, C, e D), e, posteriormente, distinto asiática dos seus homólogos com a adição de um número (A2, B2, C1, D1) . Estes nomes referem-se a quatro linhagens fundadoras que são encontrados em quase todos os do Norte, Central e do Sul populações Indígenas da América. Um pequeno número de linhagens mais raras foram recentemente identificados, mas sua contribuição para o total genética moderna Americana Nativa é provável que se mantenha o mínimo. No entanto, a conquista Européia apertou população Nativa Americana através de um gargalo genético, reduzindo conjuntos de genes Nativos Americanos em 1/3 a 1/25 de sua mudança de tamanho anterior.Esta dramática redução significativa variabilidade genética, e mudou para sempre grupos genômicos de sobreviventes. Tentativas de reconstruir as estruturas genômicas da maioria dos grupos do Novo Mundo foram recebidos com várias dificuldades decorrentes desta falta de variabilidade.
Análise genética de populações Nativas Americanas mostra que a coalescência disponível Haplogrupo gama vezes de 16.000 para 22.000 anos atrás, de acordo com a ponte de gelo hipótese.
Afiliação Native American Haplogroup e Testes
Embora um número crescente de laboratórios que oferecem testes de DNA são testes de DNA para cromossomo Y do Nativo Americano e de DNA Mitocondrial, é importante notar que estes resultados não são reconhecidos atualmente como prova suficiente para se tornar um Americano Nativo registrado. Além disso, devido ao fato de que cada um dos quatro Haplogrupos do DNA Mitocondrial principais encontrados em diferentes Grupos Tribais da América do Norte, América Central e América do Sul, um teste de DNA Nativo Americano não irá fornecer uma filiação específica Tribal.
Membros famosos Nativos Americanos Haplogroups
The Ice Maiden "Juanita" foi descoberto no Monte do Peru Ampato, perto de Arequipa, no Peru em 1995 por Johann Reinhard. "Juanita", que viveu há cerca de 500 anos atrás, foi sacrificado em uma Cerimônia Religiosa em torno da idade de 14 anos. Os Cientistas recuperaram o DNA da Ice Maiden e determinou que pertencia ao Americano Nativo Haplogrupo A2, e ela estava intimamente relacionado com a Tribo Indígena Panamenho Ngobe.
Referências
Torroni A, Schurr TG, et al. Afinidades Asiáticos e radiação continental dos quatro Fundadores Americanos Nativos. J. MtDNAs Genética Humana (1993)
Torroni A, Chen YS, et al. mtDNA e polimorfismos do cromossomo Y em quatro povos Indígenas da México.Am sul. J. Human Genetics (1994).
Bandelt HJ, Herrnstadt C., et al. Identificação dos nativos mtDNAs Fundador americano através da análise de sequências de DNA mitocondrial completas:. Algumas ressalvas Annals of Human Genetics (2003)
Forster P, R Harding, Torroni A, e Bandelt Origem e evolução da HJ mDNA variação Nativo Americano:. A reavaliaçãoAm. J. Genética Humana (1996)
Derenko MV, Grzybowski T, et al. Diversidade de linhagens de DNA mitocondrial no Sul da Sibéria. Annals of Human Genetics (2003)
Michael Crawford. As origens dos Nativos Americanos: Evidências do Genética Antropológico, Cambridge University Press (1998)
A haplogrupo é acreditado para ter originado na Ásia cerca de 60.000 anos antes do presente. Sua Haplogrupo ancestral era Haplogroup N.
A Haplogrupo é encontrado em toda a Ásia moderna. Seu subgrupo A1 é encontrada no norte e da Ásia Central, enquanto o seu subgrupo A2 é encontrado na Sibéria e é também um dos cinco Haplogrupos encontrados nos povos Indígenas das Américas, sendo os outros B, C, D e X.
A múmia "Juanita", do Peru, também chamado de "Ice Maiden", tem sido demonstrado que pertencem ao Haplogrupo Mitocondrial A.
Em seu famoso livro As Sete Filhas de Eva, Bryan Sykes nomeado o autor deste Haplogrupo Mitocondrial Aiyana.
Norte e América do Sul são os continentes mais recentemente colonizado por humanos modernos. A migração modelo a hipótese de que até o final da última Idade do Gelo (cerca de 25.000 anos atrás) um pequeno número de caçadores e coletores seguiu os padrões de migração de animais de grande porte do norte da Ásia para a América do Norte através da ponte de gelo do Estreito de Bering. Essa expansão ocorreu de forma rápida, e empurrou para os climas mais quentes da América Central e do Sul, devido às condições climáticas adversas e de gelo que cobria a maior parte da América do Norte, é provável que pequenos grupos de indivíduos permaneceram espalhados Alasca atual, em seguida, mudou para o sul em uma segunda onda , como as condições climáticas melhoraram gradualmente.
As primeiras quatro letras do alfabeto são usados para designar as quatro principais mtDNA Haplogrupos Nativos Americanos (A, B, C, e D), e, posteriormente, distinto asiática dos seus homólogos com a adição de um número (A2, B2, C1, D1) . Estes nomes referem-se a quatro linhagens fundadoras que são encontrados em quase todos os do Norte, Central e do Sul populações Indígenas da América. Um pequeno número de linhagens mais raras foram recentemente identificados, mas sua contribuição para o total genética moderna Americana Nativa é provável que se mantenha o mínimo. No entanto, a conquista Européia apertou população Nativa Americana através de um gargalo genético, reduzindo conjuntos de genes Nativos Americanos em 1/3 a 1/25 de sua mudança de tamanho anterior.Esta dramática redução significativa variabilidade genética, e mudou para sempre grupos genômicos de sobreviventes. Tentativas de reconstruir as estruturas genômicas da maioria dos grupos do Novo Mundo foram recebidos com várias dificuldades decorrentes desta falta de variabilidade.
Análise genética de populações Nativas Americanas mostra que a coalescência disponível Haplogrupo gama vezes de 16.000 para 22.000 anos atrás, de acordo com a ponte de gelo hipótese.
Afiliação Native American Haplogroup e Testes
Embora um número crescente de laboratórios que oferecem testes de DNA são testes de DNA para cromossomo Y do Nativo Americano e de DNA Mitocondrial, é importante notar que estes resultados não são reconhecidos atualmente como prova suficiente para se tornar um Americano Nativo registrado. Além disso, devido ao fato de que cada um dos quatro Haplogrupos do DNA Mitocondrial principais encontrados em diferentes Grupos Tribais da América do Norte, América Central e América do Sul, um teste de DNA Nativo Americano não irá fornecer uma filiação específica Tribal.
Membros famosos Nativos Americanos Haplogroups
The Ice Maiden "Juanita" foi descoberto no Monte do Peru Ampato, perto de Arequipa, no Peru em 1995 por Johann Reinhard. "Juanita", que viveu há cerca de 500 anos atrás, foi sacrificado em uma Cerimônia Religiosa em torno da idade de 14 anos. Os Cientistas recuperaram o DNA da Ice Maiden e determinou que pertencia ao Americano Nativo Haplogrupo A2, e ela estava intimamente relacionado com a Tribo Indígena Panamenho Ngobe.
Referências
Torroni A, Schurr TG, et al. Afinidades Asiáticos e radiação continental dos quatro Fundadores Americanos Nativos. J. MtDNAs Genética Humana (1993)
Torroni A, Chen YS, et al. mtDNA e polimorfismos do cromossomo Y em quatro povos Indígenas da México.Am sul. J. Human Genetics (1994).
Bandelt HJ, Herrnstadt C., et al. Identificação dos nativos mtDNAs Fundador americano através da análise de sequências de DNA mitocondrial completas:. Algumas ressalvas Annals of Human Genetics (2003)
Forster P, R Harding, Torroni A, e Bandelt Origem e evolução da HJ mDNA variação Nativo Americano:. A reavaliaçãoAm. J. Genética Humana (1996)
Derenko MV, Grzybowski T, et al. Diversidade de linhagens de DNA mitocondrial no Sul da Sibéria. Annals of Human Genetics (2003)
Michael Crawford. As origens dos Nativos Americanos: Evidências do Genética Antropológico, Cambridge University Press (1998)
http://www.genetree.com/a_and_b
Clã Aiyana é um dos 36 grupos de mtDNA (ou clãs) identificados até agora. De 26 de agosto
2003, Oxford Ancestors banco de dados pesquisável lista 39 jogos para Aiyana.
De acordo com a Oxford Ancestors:
Aiyana
... o fundador de um dos quatro grandes clãs que colonizaram a América do Norte e do Sul da Ásia aproximadamente 12.000 anos atrás. Descendentes Aiyana lutou extremo frio e gelo como eles cruzaram o estreito de Bering, que ligava o que é agora o Alasca ea Sibéria, para alcançar as Grandes Planícies da América do Norte. De lá, se espalhou para chegar a todas as partes do continente e, dentro de apenas alguns milhares de anos, toda a América Central e América do Sul também. No entanto, com base nos dados actualmente disponíveis, não é possível determinar com exactidão onde viviam Aiyana ou mesmo quando.
Os descendentes de quatro clãs - Chochmingwu, Djigonasee, Aiyana e Ina - dominam os Nativos Americanos. Além disso, cerca de 1% de Americanos nativos são encontrados pertencer ao clã de Xenia, que teve suas origens nas fronteiras da Europa e da Ásia. O Chochminwu clãs, Djigonasee e Aiyana pode ser encontrada em habitantes modernos da Sibéria e Alasca, mas este não é o caso de Ina. Seus descendentes são encontrados na América Central e do Sul, mas apenas para o norte até Ilha de Vancouver, no noroeste da costa do Pacífico. Curiosamente, este é também o mesmo clã que está intimamente associado com a liquidação das ilhas polinésias do sudeste da Ásia.
Os Nativos do Extremo Oriente [Ásia] são predominantemente descendentes de Djigonasee, mas não há representação considerável de "Americanos Nativos" e Ina Aiyana clãs, clãs e mais seis - Fufei, Yumi, Nene, Malaxshmi, Emiko e Gaia.
Segundo GeneTree: História Cultural de mtDNA haplogrupo A: A origem de seu Haplótipo de mtDNA remonta até 25.000 anos antes do presente, e coincide com o povoamento inicial das Américas. O povoamento das Américas ocorreu com a passagem de caçadores nômades da Sibéria para o Alasca Nordeste da Ásia pelo Estreito de Bering. Atualmente, mtDNA e cromossomo Y evidência sugere que os migrantes iniciais para as Américas teve origem no Lago Baikal / Mongólia norte. Esses nômades foram grandes caçadores extremamente móveis e rapidamente colonizaram o Novo Mundo se espalhando desde o Alasca até o extremo sul da Argentina, em apenas alguns milhares de anos. A densidade populacional aumentou e tornou-se o jogo raro grande, estes caçadores começou a diversificar a sua dieta e preencher todos os nichos ecológicos disponíveis. Na época do contato Europeu, Nativos Americanos viviam estilos de vida extremamente variadas, com alguns grupos, como no México, o desenvolvimento de complexos de base agrícola sociedades e de outros grupos, tais como a Grande Bacia, permanecendo caçadores-coletores, que vivem em pequenos grupos móveis .
Clã Aiyana é um dos 36 grupos de mtDNA (ou clãs) identificados até agora. De 26 de agosto
2003, Oxford Ancestors banco de dados pesquisável lista 39 jogos para Aiyana.
De acordo com a Oxford Ancestors:
Aiyana
... o fundador de um dos quatro grandes clãs que colonizaram a América do Norte e do Sul da Ásia aproximadamente 12.000 anos atrás. Descendentes Aiyana lutou extremo frio e gelo como eles cruzaram o estreito de Bering, que ligava o que é agora o Alasca ea Sibéria, para alcançar as Grandes Planícies da América do Norte. De lá, se espalhou para chegar a todas as partes do continente e, dentro de apenas alguns milhares de anos, toda a América Central e América do Sul também. No entanto, com base nos dados actualmente disponíveis, não é possível determinar com exactidão onde viviam Aiyana ou mesmo quando.
Os descendentes de quatro clãs - Chochmingwu, Djigonasee, Aiyana e Ina - dominam os Nativos Americanos. Além disso, cerca de 1% de Americanos nativos são encontrados pertencer ao clã de Xenia, que teve suas origens nas fronteiras da Europa e da Ásia. O Chochminwu clãs, Djigonasee e Aiyana pode ser encontrada em habitantes modernos da Sibéria e Alasca, mas este não é o caso de Ina. Seus descendentes são encontrados na América Central e do Sul, mas apenas para o norte até Ilha de Vancouver, no noroeste da costa do Pacífico. Curiosamente, este é também o mesmo clã que está intimamente associado com a liquidação das ilhas polinésias do sudeste da Ásia.
Os Nativos do Extremo Oriente [Ásia] são predominantemente descendentes de Djigonasee, mas não há representação considerável de "Americanos Nativos" e Ina Aiyana clãs, clãs e mais seis - Fufei, Yumi, Nene, Malaxshmi, Emiko e Gaia.
Segundo GeneTree: História Cultural de mtDNA haplogrupo A: A origem de seu Haplótipo de mtDNA remonta até 25.000 anos antes do presente, e coincide com o povoamento inicial das Américas. O povoamento das Américas ocorreu com a passagem de caçadores nômades da Sibéria para o Alasca Nordeste da Ásia pelo Estreito de Bering. Atualmente, mtDNA e cromossomo Y evidência sugere que os migrantes iniciais para as Américas teve origem no Lago Baikal / Mongólia norte. Esses nômades foram grandes caçadores extremamente móveis e rapidamente colonizaram o Novo Mundo se espalhando desde o Alasca até o extremo sul da Argentina, em apenas alguns milhares de anos. A densidade populacional aumentou e tornou-se o jogo raro grande, estes caçadores começou a diversificar a sua dieta e preencher todos os nichos ecológicos disponíveis. Na época do contato Europeu, Nativos Americanos viviam estilos de vida extremamente variadas, com alguns grupos, como no México, o desenvolvimento de complexos de base agrícola sociedades e de outros grupos, tais como a Grande Bacia, permanecendo caçadores-coletores, que vivem em pequenos grupos móveis .
História genética dos povos indígenas das Américas
Origem: Wikipédia, a enciclopédia livre
Para uma introdução não técnica à genética em geral, consulte Introdução à genética .

Um autossômica árvore genética mostrando algumas relações neighbor-joining dentro povos ameríndios
História genética dos povos indígenas das Américas se concentram principalmente em humanos haplogrupos de DNA do cromossomo Y e haplogrupos de DNA mitocondrial Humanos . [ 1 ] autossômicas "atDNA" marcadores também são usados, mas diferem de mtDNA ou Y-DNA em que eles se sobrepõem de forma significativa. [ 2 ] O padrão genético indicaameríndios experimentou dois episódios genéticas muito distintas; primeiro com o povoamento inicial das Américas , e em segundo lugar, com a colonização européia das Américas . [ 3 ] [ 4 ] O primeiro é o fator determinante para o número degenes linhagens, zigosidade mutações e fundadores haplótipos presentes nos atuais populações ameríndias Indígenas . [ 3 ]
A ocupação humana do Novo Mundo ocorreu em etapas a partir da linha do mar de Bering costa , com uma parada inicial naBeríngia para a pequena população de fundação . [ 5 ] [ 6 ] [ 7 ] O micro-satélite a diversidade ea distribuição da linhagem Y específicas para América do Sul indica que certas populações ameríndias foram isolados desde a colonização inicial da região. [ 8 ] O Na-Dené , Inuit e do Alasca Indígenas populações exibem haplogrupo Q (Y-DNA) ; no entanto, eles são diferentes de outros ameríndios com várias mutações do mtDNA e atDNA. [ 9 ] [ 10 ] [ 11 ] Isto sugere que os povos que se estabeleceram os extremos norte da América do Norte e Groenlândia derivada de populações migrantes posteriores do que aqueles que penetrou mais ao sul das Américas. [ 12 ] [ 13 ] Os lingüistas e os biólogos chegaram a uma conclusão semelhante com base na análise dos grupos de línguas ameríndias e do sistema ABO de grupos sanguíneos distribuições.[ 14 ] [ 15 ] [ 16 ]
Conteúdo
[ hide ]
Fundo [ editar ]
Mais informações: Cronologia da evolução humana e origem Africano recentes dos humanos modernos

Um polimorfismo de um único nucleótido (SNP) é uma alteração de um único nucleótido na sequência do ADN.
O cromossomo Y é transmitido diretamente de pai para filho; todos os seres humanos do sexo masculino (cromossomos Y) hoje remontam a um pai solteiro pré-histórico denominado " Y cromossômica Adam "originários da África. [ 17 ] O cromossomo Y se estende por cerca de 60 milhões de pares de base (os blocos de construção de DNA ) e representa cerca de 2 por cento do o DNA total em todas as células humanas . [ 18 ] O original "cromossômica Y Adam"-DNA sequenciamento sofreu uma mutação raramente ao longo dos 20 mil gerações, mas cada vez que uma nova mutação ocorre, há uma nova filial em um haplogrupo, resultando em um novo subclado ( polimorfismo de nucleotídeo único (SNP)). [ 19 ]
Mutações do mtDNA também são passadas relativamente inalterados de geração em geração, de modo que todos os seres humanos compartilham os mesmos tipos de mtDNA. A extensão lógica disso é que todos os seres humanos, em última análise remontam a uma mulher, que é comumente referido como a Eva Mitocondrial . [ 20 ] [ 21 ] Machos e fêmeas herdam o DNA mitocondrial (mtDNA), apenas a partir de sua mãe. [ 22 ] Esta linha de herança biológica, portanto, para com cada homem. [ 23 ] Consequentemente, Y-DNA é mais comumente utilizado pelo público em geral para o rastreamento do patrimônio genético de uma linhagem masculina direta. [ 23 ] [ 24 ] [ 25 ]
Um autosome (atDNA) é um cromossoma que não é um cromossoma sexual - isto é, existem um igual número de cópias do cromossoma em machos e fêmeas. [ 2 ] um teste de DNA autossómica é geralmente usada para determinar as percentagens genéticas " "de ancestralidade de uma pessoa a partir de determinado continentes / regiões ou para identificar os países e as" tribos "de origem em uma base global.miscigenação genética testes chegar a esses percentuais, examinando (SNP), que são locais no DNA, onde um nucleotídeo tem " mutante "ou" ligado "a um diferente nucleotídeo . [ 2 ] Uma forma de examinar o suporte para determinadas rotas de colonização dentro da massa de terra norte-americana é para determinar se uma relação mais estreita entre zigosidade e geografia é observado quando "eficazes" as distâncias geográficas são computados juntamente estas rotas, em vez de ao longo de caminhos de menor distância. [ 26 ]
Y-DNA [ editar ]
- Para mais detalhes sobre os grupos indígenas individuais por Y-DNA, ver haplogrupos Y-DNA em povos indígenas das Américas .
O cromossomo Y consórcio estabeleceu um sistema de definição de haplogrupos Y-DNA por letras de A a T , com subdivisões usando números e letras minúsculas. [ 27 ]
Haplogrupo Q [ editar ]
Para mais detalhes sobre este tópico, consulte haplogrupo Q-M242 (Y-DNA) .
Q-M242 (nome mutacional) é a definição (SNP) do haplogrupo Q (Y-DNA) (nome filogenética). Dentro do clado Q, há 14 haplogrupos marcados por 17 SNPs. 2.009 [ 28 ] [ 29 ] Na Eurásia haplogrupo Q é encontrado entre indígenaspopulações siberianas , como os modernos Chukchi e Koryak povos. Em particular, dois grupos apresentam grandes concentrações de mutação Q-M242, a Ket (93,8%) eo Selkup (66,4%) pessoas. [ 30 ] A Ket são pensados para ser os únicos sobreviventes de antigos andarilhos que vivem na Sibéria. [ 5 ] O tamanho da população é muito pequena; há menos de 1.500 Ket em Rússia . 2.002 [ 5 ] O Selkup ter um tamanho de população pouco maior que o Ket, com cerca de 4.250 indivíduos. [ 31 ]
Iniciando o período Paleo-índios , a migração para as Américas através do Estreito de Bering ( Beríngia ) por uma pequena população portador da mutação Q-M242 ocorreu. [ 32 ] Um membro desta população inicial sofreu uma mutação, que define sua população descendente , conhecida pela Q-M3 mutação (SNP). [ 33 ] Esses descendentes migraram todas as Américas. [ 28 ]
Haplogrupo Q-M3 (Y-DNA) é definida pela presença dos rs3894 (M3) (SNP). [ 3 ] [ 5 ] [ 34 ] A mutação Q-M3 é aproximadamente 15.000 anos de idade, como é que, quando a migração inicial de paleoíndios na América ocorreu. [ 7 ] [ 35 ] Q-M3 é o haplótipo predominante nas Américas, a uma taxa de 83% nas populações da América do Sul, [ 36 ] 50% nas populações de Na-Dené, e na América do Norte populações do Esquimó-Aleut em cerca de 46%. [ 30 ] Com o mínimo de back-migração de Q-M3 na Eurásia, a mutação provavelmente evoluiu na leste-Beríngia, ou mais especificamente a Península Seward ou interior do Alasca ocidental . A massa de terra Beríngia começou submergindo, cortando rotas terrestres. [ 6 ] [ 30 ] [ 37 ]
Desde a descoberta de Q-M3, várias subclasses de populações M3 de rolamento foram descobertos. Um exemplo está na América do Sul , onde algumas populações têm uma elevada prevalência de (SNP) M19 que define subclado Q-M19 . [ 36 ] M19 foi detectado em (59%) da Amazônia Ticuna homens e (10%) de Wayuu homens. [ 36 ] subclado M19 parece ser exclusivo para os povos do Sul indígenas americanas , decorrentes de 5.000 a 10.000 anos atrás. [ 36 ] Isto sugere que o isolamento da população e talvez até mesmo a criação de grupos tribais começou logo após a migração para as áreas sul-americanos. [ 5 ] [ 38 ] Outras subclasses americanas incluem Q-L54 , Q-Z780 , Q-MEH2 , Q-SA01 , e Q-M346 linhagens. No Canadá, duas outras linhagens foram encontrados. Estes são Q-P89.1 e Q-NWT01 .
Haplogrupo R1 [ editar ]
Para mais detalhes sobre este tópico, consulte Haplogrupo R1 (Y-DNA) .
Haplogrupo R1 (Y-DNA) (especialmente R 1b ) é o segundo haplótipo Y mais predominante entre ameríndios após Q (Y-DNA). [ 39 ] A distribuição de R1 se acredita estar associada com o re-estabelecimento da sequência Eurásia o último máximo glacial . Uma teoria colocar diante é que ele entrou nas Américas com a população fundação inicial. [ 40 ] A segunda teoria é que ele foi introduzido durante a colonização européia. [ 39 ] R1 é muito comum em toda a Eurásia, exceto a Ásia Oriental e Sudeste da Ásia . R1 (M173) é encontrado predominantemente em grupos norte-americanas, como o ojíbua (79%), chipewyan (62%), Seminole (50%), Cherokee (47%), dogrib (40%) epapago (38%). [ 39 ]
Haplogrupo C-P39 [ editar ]
Para mais detalhes sobre este tópico, consulte haplogrupo C-M217 (Y-DNA) .
Haplogrupo C-M217 é encontrada principalmente em siberianos indígenas, os mongóis e as populações oceânicas. Haplogrupo C-M217 é o ramo mais difundido e frequente do maior (Y-DNA) haplogrupo C-M130 . Haplogrupo C-M217 descendente C-P39 é comumente encontrada em de hoje oradores na-dene , com a maior freqüência encontrada entre os Athabaskans em 42%. [ 32 ] Este ramo distinto e isolado C-P39 inclui quase todo o haplogrupo C-M217 Y -cromossomos encontrados entre todos os povos indígenas das Américas. [ 41 ] Os grupos na-dene também são incomuns entre os povos indígenas das Américas a ter uma freqüência relativamente alta de Q-M242 (25%). [ 30 ]
Alguns pesquisador acha que isso pode indicar que a migração Na-Dené ocorrido a partir do Extremo Oriente russo , após a colonização Paleo-índio inicial, mas antes da moderna Inuit , Inupiat e Yupik expansões. [ 9 ] [ 10 ] [ 42 ]
mtDNA [ editar ]
Para mais detalhes sobre mtDNA em geral, consulte DNA mitocondrial .
Eva Mitocondrial é definida como a mulher que foi o matrilinear ancestral comum mais recente de todos os que vivem os seres humanos . Eva mitocondrial é estimada para ter vivido entre 140.000 e 200.000 anos atrás, [ 43 ] [ 44 ] Eva mitocondrial é o mais recente comum matrilinear ancestral, e não o mais recente ancestral comum . [ 45 ] [ 46 ]
Ao estudar o DNA mitocondrial humano (mtDNA) haplogrupos , os resultados indicaram que, até recentemente, haplogrupos ameríndios Indígenas, incluindo haplogrupo X , são parte de um único fundador população do leste asiático. Ele também indica que a distribuição de haplogrupos do DNA mitocondrial e os níveis de divergência de seqüência entre os grupos lingüisticamente similares foram o resultado de múltipla anterior migrações de Bering Straits populações. [ 22 ] [ 47 ] All Indígena ameríndia mtDNA pode ser rastreada até cinco haplogrupos, A , B , C , D e X . [ 48 ] Mais especificamente, indígenas ameríndios mtDNA pertence à sub-haplogrupos que são exclusivos para as Américas e não encontrada na Ásia ou na Europa: A2, B2, C1, D1, e X2a (com menor grupos C4c, D2, D3 e D4h3). [ 47 ] Isto sugere que 95% dos indígenas ameríndios mtDNA é descendente de um fundador população feminina genética mínima, composta por sub-haplogrupos A2, B2, C1b, C1c, C1d e D1. [ 48 ] O 5% restante é constituído pelo X2a, D2, D3, C4, e D4h3 sub-haplogroups. [ 47 ] [ 48 ]
X é um dos cinco haplogrupos do mtDNA encontrados em povos ameríndios Indígenas. Ao contrário dos quatro principais haplogrupos americano mtDNA (A, B, C e D), X não é de todo fortemente associada com a Ásia oriental. [ 5 ] seqüências genéticas haplogrupo X divergiram cerca de 20.000 a 30.000 anos atrás para dar dois sub-grupos, X1 e X2. Subclado X2a do X2 só ocorre com uma frequência de cerca de 3% da população indígena total atual das Américas. [ 5 ] No entanto, é uma das principais X2a subclado mtDNA na América do Norte; entre os povos algonquinos , compreende-se a 25% dos tipos de mtDNA. [ 49 ] [ 50 ] Ele também está presente em percentagens mais baixas a oeste e ao sul desta área - entre os Sioux (15%), o Nuu-chah- nulth (11% -13%), o Navajo (7%), ea Yakama (5%). [ 51 ] haplogrupo X é mais fortemente presente no Oriente Médio , o Cáucaso ea Europa Mediterrânea . [ 51 ] A predominante teoria para a aparição de sub-haplogrupo X2a na América do Norte é a migração, juntamente com A, B, C, e D do mtDNA grupos, a partir de uma fonte nas montanhas de Altai da Ásia central. [ 52 ] [ 53 ] [ 54 ] [ 55 ]
O seqüenciamento do genoma mitocondrial de Paleo-Esquimó permanece (3.500 anos) são distintos dos índios modernos, caindo dentro de sub-haplogrupo D2A1, um grupo observado entre hoje Aleutas Islanders , o aleúte e Siberian Yupik populações. [ 56 ] Isto sugere que o colonizadores do extremo norte, e, posteriormente, Groenlândia, originado de populações costeiras posteriores. [ 56 ] Em seguida, uma troca genética nos extremos norte introduzidas pelas pessoas de Thule (proto-Inuit) cerca de 800-1.000 anos atrás começou. [ 11 ] [ 57 ] Estes imigrantes pré-colombianos finais introduziu haplogrupos A2a e A2b às populações Paleo-Eskimo existentes do Canadá e da Groenlândia, que culminou com o moderno Inuit . [ 11 ] [ 57 ]
Um estudo de 2013, em Nature informou que o DNA encontrado nos 24.000 anos de idade, restos de um jovem rapaz da cultura Mal'ta-Buret 'arqueológico sugerem que até um terço dos norte-americanos podem ter ascendência indígena que pode ser rastreada para Eurasians ocidentais , que podem ter "tiveram uma distribuição mais nordeste 24.000 anos atrás do que normalmente se pensa" [ 58 ] "Nós estimamos que 14 a 38 por cento de ascendência americana nativa pode se originar através de fluxo gênico dessa população antiga," os autores escreveu. Professor Kelly Graf disse:
"Nossos resultados são significativos em dois níveis. Primeiro, ele mostra que Paleolítico Superior siberianos veio de uma população cosmopolita de humanos modernos que se espalhou para fora da África para a Europa Central e Sul da Ásia. Segundo, esqueletos Paleoindian como Buhl mulher com traços fenotípicos atípico da moderna indígenas americanos pode ser explicada como tendo uma ligação histórica direta ao Paleolítico Superior Sibéria ".
A rota através Beríngia é visto como mais provável do que a hipótese Solutrean . [ 59 ] Um resumo em uma edição de 2012 do "American Journal of Physical Anthropology", afirma que "As semelhanças nas idades e distribuições geográficas para C4c ea linhagem X2a previamente analisadas fornecer suporte ao cenário de uma dupla origem para Paleo-índios. Tendo em conta que C4c está profundamente enraizado na parte asiática da filogenia mtDNA e é indubitavelmente de origem asiática, a constatação de que C4c e X2a são caracterizadas por histórias genéticas paralelas definitivamente descarta a hipótese controversa de uma rota de entrada glacial Atlântico na América do Norte ". [ 60 ]
AtDNA [ editar ]
Para mais detalhes sobre DNA autossômico em geral, consulte a variação genética humana .

grupos genéticos de acordo com Tribos de DNA
1. Ártico
2. Salishan
3. Athabaskan
4. Norte ameríndia
5. Ojibwa
6. Mexica
|
7. Maya
9. Andina
10. Amazônia
11. Gran Chaco
12. Patagônia
|
Diversidade genética e estrutura populacional em massa de terra norte-americana também é feito usando autossômico (atDNA) marcadores micro-satélites genotipados ; amostrados a partir de Norte, Central e América do Sul e analisados em relação aos dados similares disponíveis de outras populações indígenas em todo o mundo. [ 26 ] [ 61 ] As populações ameríndias mostram uma menordiversidade genética de populações de outras regiões continentais. [ 61 ] Observado é uma genética diminuindo diversidade como distância geográfica do Estreito de Bering, ocorre, bem como uma semelhança genética para diminuir as populações da Sibéria do Alasca (o ponto de entrada genética). [ 26 ] [ 61 ]
Também observou evidência de um maior nível de diversidade e menor nível de estrutura populacional no oeste da América do Sul em relação ao Leste da América do Sul. [ 26 ] [ 61 ] Há uma relativa falta de diferenciação entre mesoamericana e andina populações, um cenário que implica que rotas costeiras eram mais fáceis para a migração de povos (contribuintes mais genéticas) para atravessar em comparação com rotas terrestres. [ 26 ]
O padrão de sobre-tudo que está surgindo sugere que as Américas foram colonizadas por um pequeno número de indivíduos (tamanho efetivo de cerca de 70), que cresceu por um fator de 10, mais de 800 -. Mil anos [ 5 ] [ 62 ] Os dados também mostra que houve trocas genéticas entre a Ásia, o Ártico ea Groenlândia desde o povoamento inicial das Américas. [ 62 ] [ 63 ]
Em 2014, a DNA autossômico de um bebê + 12.500 anos de idade, a partir de Montana foi sequenciado. [ 64 ] O DNA foi extraído de um esqueleto chamado de Anzick-1, encontrados em estreita associação com vários artefatos Clovis. As comparações mostraram afinidades fortes com o DNA a partir de sites da Sibéria, e praticamente descartou qualquer afinidade com fontes europeias (a chamada " hipótese Solutrean "). O DNA também mostrou fortes afinidades com todas as populações nativas americanas existentes, o que indica que todos eles derivam de uma população ancestral que viveu em ou perto da Sibéria, o Paleolítico Superior Mal'tapopulação. [ 65 ]
Sobreposições entre os tipos de DNA [ editar ]
Mais informações: Genética de populações e haplogrupos Y-DNA em povos indígenas das Américas
As populações que têm uma combinação específica de autosome, Y e mutações MT-haplogroup pode geralmente ser encontrada com variações regionais. Autossomos, mutações Y e mutações mt não ocorrem necessariamente em um momento semelhante e há taxas diferenciadas de seleção sexual entre os dois cromossomos sexuais. [ 66 ] Isto combinado com gargalos populacionais , oefeito fundador , mutações mitocondriais e deriva genética irá alterar a composição genética das populações isoladas, resultando em padrões de mutação muito distintas. [ 67 ](ou seja, Tainos , [ 68 ] Fuegians , [ 69 ] Inuit , [ 70 ] Yupik [ 71 ] e Algonquian [ 5 ] )
As sobreposições ásperas entre Y-DNA e DNA mitocondrial entre as Américas, norte Circumpolar , e as populações indígenas da Sibéria são:
| Haplogrupo (s) Y-DNA - | haplogrupo (s) mtDNA - | Área geográfica (s) |
|---|---|---|
| Q-M242 , R1 , C-M217 | A , X , Y , C , D ( M tipos), ( N tipos) | Extremo Oriente russo , Américas , Ártico |
Mistura genética do Velho Mundo [ editar ]
Para mais detalhes sobre mistura genética nas Américas, ver estudos Miscigenação § genéticos da mistura racial .
Veja também: multiétnico americano

"Las castas" - Pintura contendo conjunto completo de 16 Casta combinações.Um sistema de classificação sócio-racial do século 18 usado nas colônias hispano-americanas.
Casamento inter-racial e inter-racial sexo e, mais geralmente, o processo de mistura racial , tem suas origens na pré-história. Mistura racial tornou-se comum durante Europeia colonialismo na Era dos Descobrimentos . [ 72 ] troca genética entre duas populações reduz a distância genética entre as populações e é mensurável no DNA padrões. [ 66 ] Durante a época dos Descobrimentos, com início no final dos anos 1400, exploradores europeus navegaram os oceanos, eventualmente, chegar a todos os principais continentes. [ 73 ] Durante este tempo, os europeus contactado muitas populações, alguns dos quais tinham sido relativamente isoladas por milênios . [ 74 ] A composição demográfica genética do Hemisfério Oriental não mudou significativamente desde a idade da descoberta. No entanto, a demografia genéticos no Hemisfério Ocidental foram radicalmente alterados por eventos seguintes as viagens de Cristóvão Colombo . [ 74 ] A colonização européia das Américas trouxe o contato entre os povos da Europa, África e Ásia e as populações ameríndias. Como resultado, as Américas têm hoje significativos e complexosmultirraciais populações. [ 74 ] Muitas pessoas que se auto-identificam como uma raça apresentam evidência genética de uma ascendência multirracial . [ 75 ]
A conquista européia da América Latina a partir do final do século 15, foi inicialmente executado por soldados do sexo masculino e marinheiros da Península Ibérica ( Espanha e Portugal ). [ 76 ] Os novos soldados-colonos teve filhos com mulheres indígenas e, posteriormente, com os escravos africanos . [ 77 ] Estes mestiços crianças eram geralmente identificado pelo colonizador espanhol e colono Português como " Castas " .[ 78 ] A subsequente comércio de peles norte-americano durante o século 16 trouxe muitos mais homens europeus, de França , Irlanda e Grã-Bretanha , que tomou mulheres norte-ameríndias como esposas. [ 79 ] Seus filhos se tornaram conhecidos como " Métis " ou " Bois-brules " peloscolonos franceses e " mestiços " , " mestiços " ou " nascido de país " por parte do colonos ingleses e colonos escoceses . [ 80 ] A partir da segunda metade do século 19 até o início do século 20, novas ondas de imigrantes da Europa do norte, leste e sul foram para as Américas e, conseqüentemente, alterou os dados demográficos . [ 81 ] Na sequência II Guerra Mundial e as migrações posteriores em todo o mundo, os atuaispopulações americanas mistura genética 'pode ser atribuída a todos os cantos do mundo. [ 5 ] [ 82 ]
Grupos sanguíneos [ editar ]
Mais informações: sistema de grupo sanguíneo ABO
Antes de 1952 a confirmação do DNA como o material hereditário por Alfred Hershey e Martha perseguição , os cientistas usaram proteínas do sangue para estudar a variação genética humana. [ 83 ] [ 84 ] O sistema de grupo sanguíneo ABO é creditado por ter sido descoberto pelo austríaco Karl Landsteiner , que encontrou três tipos diferentes de sangue em 1900. [ 85 ] Grupos sanguíneos são herdadas de ambos os pais. O tipo de sangue ABO é controlada por um único gene (o gene ABO ) com três alelos : i , I A , e I B . [ 86 ]
Uma pesquisa feita por Ludwik e Hanka Herschfeld durante a I Guerra Mundial descobriram que as freqüências degrupos sanguíneos A, B e O diferia muito de região para região. [ 84 ] O "o" tipo de sangue (normalmente resultante da ausência de ambos os alelos A e B ) é muito comum em todo o mundo, com uma taxa de 63% em todas as populações humanas. [ 87 ] Tipo "O" é o tipo de sangue principal entre as populações indígenas das Américas, em particular dentro de populações da América Central e do Sul, com uma freqüência de quase 100%. [ 87 ] Em populações indígenas da América do Norte a freqüência de tipo "A" varia de 16% a 82%. [ 87 ] Isto sugere novamente que os ameríndios iniciais evoluíram a partir de uma população isolada com um número mínimo de indivíduos. [ 88 ] [ 89 ]
| PEOPLE GROUP | O (%) | A (%) | B (%) | AB (%) |
|---|---|---|---|---|
| Blackfoot (N. Am. indiano) | 17 | 82 | 0 | 1 |
| Bororo (Brasil) | 100 | 0 | 0 | 0 |
| Esquimós (Alasca) | 38 | 44 | 13 | 5 |
| Inuit (leste do Canadá e Groenlândia) | 54 | 36 | 23 | 8 |
| Havaianos | 37 | 61 | 2 | 1 |
| Norte-americanos Indígena (como um todo Unidas Native / Primeiras Nações ) | 79 | 16 | 4 | 1 |
| Maya (moderna) | 98 | 1 | 1 | 1 |
| Navajo | 73 | 27 | 0 | 0 |
| Peru | 100 | 0 | 0 | 0 |
Teste Genealógico [ editar ]
: Ver artigo principal teste genealógico do ADN
Um teste de DNA genealógica examina os nucleotídeos em localizações específicas de uma pessoa DNA para genealogia genética propósitos. [ 91 ] Os resultados dos testes não são feitos para ter qualquer valor medicinal; eles são destinados apenas para dar informações genealógicas. Testes de DNA genealógicos geralmente envolvem a comparação dos resultados de indivíduos que vivem as populações históricas. [ 91 ] O procedimento geral para fazer um teste de DNA genealógica envolve tomar uma indolor (também conhecido como um cotonete bucal) em casa e enviá-raspagem bochecha a amostra para um laboratório genealogia genética para o teste. Resultados de testes genéticos que mostram específico sub-Haplogroups de Q , R1 e C3b implica que a ascendência indivíduos é, no todo ou em parte, originária das Américas. [ 92 ] Se alguém do mtDNA pertencia a determinado sub-Haplogroups de, A , B , C , D ou X2a , a implicação seria que a ascendência do indivíduo é, no todo ou em parte, originária das Américas. [ 93 ]
Veja também [ editar ]
Postado por ANTONIO FLORENTINO como 2012/12/02 12:21:00











Nenhum comentário:
Postar um comentário