- Meu Haplogroup mtDNA L2a1c1
Meu Haplofroup mtDNA L2a1c1 é uma das Fundadoras do povo Judeu Matriarcal Azquenazi,
Meu Haplogroup mtDNA L2a1c1 tem uma origem Norte Africano. É definido por marcadores de 198, 930, 3308, 8604, 16086. Observa-se entre a Tunísia Sefardita, Ashkenazi, Judeus, Marroquinos, Egípcios, Núbios,Hebreus e Yemenitas. http://en.wikipedia.org/wiki/Haplogroup_L2_ (mtDNA)
Meu Haplogroup mtDNA L2a1c'1 -
L2a1c1 Mutações
HVR1 diferenças rCRS 16086C 16223T 16278T 16294T 16309G 16390A
HVR2 Diferenças rCRS 73G 195C 198T 152C 146C 143A 263G 315.1C
Região Diferenças codificação de rCRS 750g 769A 930A 1018A 3010A 1438G 2706G
4104G 2416C 2789T 4769G 6663G 3308C 3594T 6164T 6311T 7028T 7256T 7274T
7771G 8188G 8701G 8860G 7175C 7521A 9221G 8206A 8604C 9540C 10115C 10873C
11137C 11719A 11914A 13590A 10398G 12693G 11944C 12705T 13650T 14766T 13803G
14566G 15326G 15301A 15784C
Haplogrupo - L2a1c1
Minha origem
Parte do conjunto L de haplogrupos, que foi concretamente caracterizados como representando a linhagem mitocondrial humana original, haplogroup L2a é encontrado na África. Este datas haplogroup a aproximadamente 55.000 anos atrás, e é detectado em maior freqüência, no norte, oeste e África central. O trabalho futuro irá documentar ainda mais a distribuição histórica deste haplogrupo e haplogrupos intimamente relacionado ao conjunto L.
Política de Uso: Uso da descrição Haplogrupo acima requer permissão por escrito da Gene por Gene.
Meus Resultados
DIFERENÇAS DE HVR1 rCRS
|
DIFERENÇAS DE HVR2 rCRS
|
CODIFICAÇÃO DIFERENÇAS região a partir rCRS
|
Revisado Cambridge seqüência de referência
Haplogroup mtDNA L2a1c1a - Resultados RSRS
Minha origem
Parte do conjunto L de haplogrupos, que foi concretamente
caracterizados como representando a linhagem mitocondrial
humana original, haplogroup L2a é encontrado na África.
Este datas haplogroup a aproximadamente 55.000 anos
atrás, e é detectado em maior freqüência, no norte, oeste e
África central.
O trabalho futuro irá documentar ainda mais a distribuição
histórica deste haplogrupo e haplogrupos intimamente
relacionado ao conjunto L.
Política de Uso: Uso da descrição Haplogrupo acima requer
permissão por escrito da Gene por Gene.
Meus Resultados
| Mutações extra |
315.1C
522.1A
522.2C
C6164T
A8188G
T11137C
C16519T
|
|---|---|
| Mutações desaparecidas |
DIFERENÇAS DE HVR1 RSRS
|
DIFERENÇAS DE HVR2 RSRS
|
CODIFICAÇÃO DIFERENÇAS região a partir RSRS
|
https://www.familytreedna.com/public/archaeologistDNA/default.aspx?vgroup=archaeologistDNA&vgroup=archaeologistDNA&vgroup=archaeologistDNA&vgroup=archaeologistDNA&vgroup=archaeologistDNA&vgroup=archaeologistDNA&vgroup=archaeologistDNA§ion=mtresults
Arqueólogo Projeto DNA - mtDNA resultados de teste para membros
A tabela abaixo mostra cada projeto memberâ número kit € ™ s e seu DNA mitocondrial testada (mtDNA) resultados. De acordo com as definições do ™ € projectâ s, ele também pode exibir o antepassado mais distante conhecido de cada membro do projeto. Esta seção apresenta os resultados do mtDNA como diferenças da histórica revista Cambridge Reference Sequence (rCRS). O rCRS é desde a primeira sequência completa de mtDNA concluída. As informações contidas nesta seção é apenas para referência histórica.
Você pode saber mais sobre o mtDNA nos resultados mtDNA Compreender página aprendizagem.
Meu Haplogroup mtDNA Arqueológico RSRS
https://www.familytreedna.com/public/archaeologistDNA/default.aspx?vgroup=archaeologistDNA&vgroup=archaeologistDNA&vgroup=archaeologistDNA&vgroup=archaeologistDNA&vgroup=archaeologistDNA&vgroup=archaeologistDNA§ion=mtresults
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Projetos Judeus Ashkenazi
Veja o projeto principal em:
Veja o projeto principal em:
Mães Fundadora Haplogroup mtDNA Judeus Ashkenazi
Mães para mais informações.
Projetos Mães Fundadora Judeus Ashkenazi
Veja o projeto principal em Fundação DNA Judeus Ashkenazi
Mães para mais informações.
Projetos Mães Fundadora Judeus Ashkenazi
Veja o projeto principal em Fundação DNA Judeus Ashkenazi
Mães para mais informações.
- HV1 (mtDNA) (Ashkenazi)
- H * (mtDNA) (Ashkenazi)
- J (mtDNA) (Ashkenazi)
- K1a1b1a (mtDNA) (Ashkenazi)
- L2a1c1 (mtDNA) (Ashkenazi)
- R0a2 (mtDNA) (Ashkenazi)
- N1b2 (mtDNA) (Ashkenazi)
- N1C (mtDNA) (Ashkenazi)
- U1B (mtDNA) (Ashkenazi)
- U4 (mtDNA) (Ashkenazi)
- T2 (mtDNA) (Ashkenzai & Sephardi)
- V (mtDNA) (Ashkenazi)
Minha Mãe mtDNA Haplogroup L2a1c1 é observada entre os Norte-Africanos, Árabes,Judeus Sefarditas Tunísianos, Judeus Ashkenazi, Hebreus, Egípcios, Núbios, Yemenitas, Marroquinos, Península Ibérica (Espanha e Portugal) Angola Português, Mbundu Angola Tribo.
Minha Mãe tem um mtDNA Haplogroup L2a1c1 jogo HVR1 com o Mbundu de Angola.
O Mbundu são um dos povos Bantu. eles estavam vindo para ter Região Angola, desde a Idade Média adiantada, mas a maior parte da imigração ocorreu entre os dias 13 e 16 de século, do Norte da África.
Kimbundu-Oeste é uma língua Bantu, e diz-se que o Mbundu ter vindo do Norte de África, em vez de a África Oriental para corresponder o que minha mãe mtDNA Haplogroup L2a1c1 origem norte Africano.
Eu tenho uma combinação de genética com o Ewondo Pessoas que estão relacionados à forma como as pessoas Beti-Pahuin.
A Beti-Pahuin de origens exatas não são claras.
Em um ponto, eles foram pensados para ter migrado para Território para Camarões atual e Norte da África na área do Sudão Azande Esta localização geográfica Norte Africano suporta uma atribuição de mtDNA Haplogroup Meu L2a1c1 ..
Meu mtDNA Haplogroup L2a1c1 tem relação com a genética como pessoas Ga Gana.
Alguns estudiosos acreditam que o povo Ga-Adangbe originou a leste da sua localização actual não Plains Accra.
mtDNA Haplogroup plausível com o meu L2a1c1, não tem a migração para a África Ocidental.
Minha origem Norte-Africano tem freqüências notáveis da Península Ibérica (Portugal e Espanha).
Este grupo compartilha um ancestral com o Espanhol Judeus Sefarditas e Mouros da Península Ibérica ..
Não contraditórias, ele também possui ascendência Mourisca Haplótipos entre os quais Mouros Muçulmanos que praticam o Islã privado, que eram Marranos Hebreus / Judeus Sefarditas que se converteu ao Cristianismo, mas manteve-se como Tradições Judaicas, em particular ..
A maior freqüência de L2a1c1 é encontrada em Hebreus Sudeste Asiático Iêmen e Omã ..
Localizador de Família - Finder População
DETALHES
| Continente (Subcontinente) | População | Percentagem | Margem de erro |
|---|---|---|---|
| África (Oeste Africano): | Yoruba | 77,82% | ± 0,28% |
| Oriente Médio: | Judeu, Palestino, beduíno, Beduíno do Sul, drusos | 8,11% | ± 1,97% |
| Europa: | Finlandês, Russo | 14,07% | ± 2,06% |
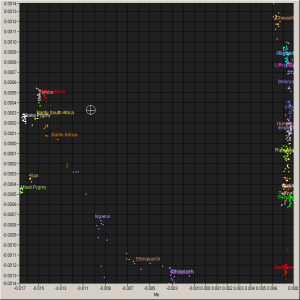
MINHAS Analises de Misturas Autossomicas:
214911-autosomal-o-results.csv.
Mais provável é ajuste de 24,9% (+ - 14,4%) África (subcontinentes diversos)
e 58,2% (+ - 15,3%) África (todos do Oeste Africano)
que é 83,1%, total de África
e 16,9% (+ - 1,2%) Europa (subcontinentes diversos)
A seguir, são conjuntos populacionais possíveis e respectivas fracções,
provavelmente no topo
Bantu Ke = 0439 = 0382 russo Mandenka = 0179 ou
Maasai = 0,152 Yoruba = 0,671 = 0,177 ou russo
O-etíope = 0,129 = 0,711 Yoruba Finlândia = 0,160 ou
O-etíope = 0,129 Yoruba = 0,710 = 0,161 ou russo
Bantu Ke = 0431 = 0391 Mandenka Finlândia s = 0178
Maasai = 0,149 Yoruba = 0,675 = 0,176 ou Finlândia
A-etíope = 0,116 Yoruba = 0,734 = 0,150 ou Finlândia
Bantu Ke = 0396 = 0424 irlandesa Mandenka = 0180 ou
T-etíope = 0,115 Yoruba = 0,734 = 0,151 ou Finlândia
Bantu Ke = 0433 = 0389 Bielorússia Mandenka = 0178
Europa ocidental, mas também é muito possível.
3,2% Native American (subtrai Europeia).
Doug
Só mais uma coisa: o cromossomo X é todo azul, ou seja, 100% Africano. Anexada.
Doug
Este foi o primeiro que eu já vi, sem uma pergunta secreta.
Um ajuste personalizado é melhor do que o automático:
Irlandesa Yoruba 0,7159 0,1461 0,0259 0,1122 O-Maya etíope ou
Irlandesa Na-Dene 0,0256 0,1467 0,7156 Yoruba de O-etíope 0,1120
Inglês Yoruba 0,7146 0,1431 0,0279 0,1144 O-Maya etíope ou
Inglês Na-Dene 0,0280 0,1435 0,7143 Yoruba de O-etíope 0,1142
Irlandesa Mandenka 0,1091 0,0255 0,1897 0,6758 Maya Yoruba
Francês ou espanhol também é possível, no mesmo montante, com a mesma probabilidade.
O-etíopes stands para Oromo, mas isso não é provavelmente real: significa apenas que o
Africano é parte de algum lugar bewteen Nigéria e Etiópia., Muito mais próxima da Nigéria.
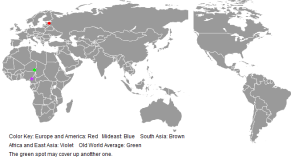
O Oriente Médio (violeta) nos Cromossomos vem do Leste Africano, eles
são misturados com pessoas de Saudita.
O Norte-Americano no Genérico Norte-Americano. A mancha vermelha (euro) em teh mapa
está no lugar errado.
Doug McDonald
Doug McDonald
Referências:
Os mouros do Norte de África e da Península Ibérica no Wiki: http://en.wikipedia.org/wiki/Moors
Os Morsicos (muçulmanos convertidos ao cristianismo) http://en.wikipedia.org/wiki/Morisco
O Murranos (hebraico converte ao cristianismo) http://en.wikipedia.org/wiki/Marrano
O Sehardim Hebreus / sefaraditas: http://en.wikipedia.org/ wiki / Sephardi_Jews
pessoas mbundu do norte http://en.wikipedia.org/wiki/Northern_Mbundu_people Camarões
do Norte Língua Mbundu: http://en.wikipedia.org/wiki/North_Mbundu_language
O Camarões Ewondo / Beti-Pahuin: http:// en.wikipedia.org / wiki / Beti-Pahuin
Haplogroup L2 Wikipedia: http://en.wikipedia.org/wiki/Haplogroup_L2_ (mtDNA)
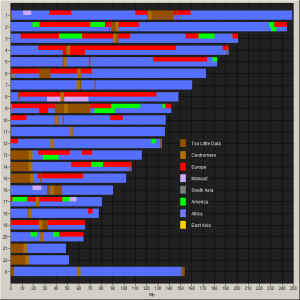
Haplogroup L2a1c1 Norte África Fonte: http://www.biomedcentral.com/1471-2164 / 9/267/figure/F2? highres = y













Nenhum comentário:
Postar um comentário